Naszym celem jest wstępne analiza graficzne przeprowadzona za pomocą Seaborn, będącą biblioteką Python.
Analiza ma odpowiedzieć na pytanie, jak niektóre wyniki pomiarów parametrów organizmu pacjentów wpływają na zapadalność lub brak zapadalności na cukrzycę.
g = sns.pairplot(data=df[['Insulin' ,'Age','SkinThickness', 'Outcome']], hue='Outcome', dropna=True, height=3, aspect=2, palette=('cubehelix'), markers='o')
g.axes[2,1].set_xlim((0,140))
g.axes[1,2].set_xlim((0,60))
Przyjrzyjmy się bliżej pobranej próbce badań.
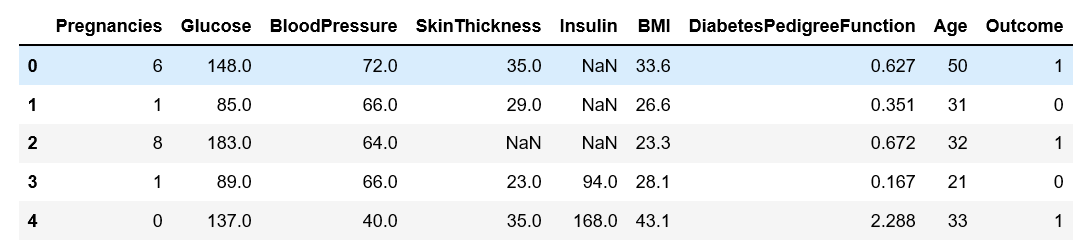
Uruchamiam najważniejsze biblioteki oraz wyświetlam 10 pierwszych wierszy bazy.
Widać, że próbki zawierają błędy. BloodPressure wykazujące wartość zero oznacza, że pacjent podczas badania już nie żył. Podobnymi błędami są brak grubości skóry czy zerowy poziom insuliny we krwi.
Próbka posiadająca tak wiele braków nie nadaje się do prowadzenia badań analitycznych. Należy więc oczyścić dane z oczywistych błędów i braków.
Python posiada wiele metod uzupełniania braku wartości, można uzupełniać braki danych średnią pomiarów lub też ostatnim dobrym pomiarem. Niestety nie będziemy stosować takich technik ponieważ tak poprawiona próbka mogłaby doprowadzić nas do nieprawdziwych wyników badań.
Na początku dowiedzmy się coś więcej o próbce.
df.dtypes
Teraz sprawdzimy ile mamy wierszy i kolumn w bazie.
df.shape
Teraz powinniśmy wykasować wiersze, które nie mają kompletu danych. Być może stracimy bardzo dużą część rekordów, jednak na końcu zyskamy czyste dane, bez braków i bez fałszywych wartości.
Sprawdzam ile jest wartości zerowych w kolumnach: ['Glucose','BloodPressure','SkinThickness', 'Insulin','BMI']
(df[['Glucose','BloodPressure','SkinThickness', 'Insulin','BMI']]==0).sum()
Teraz aby skutecznie wykasować niekompletne rekordy zamieniamy zera w tych kolumnach na wartości np.NaN
df[['Glucose','BloodPressure','SkinThickness', 'Insulin','BMI']] = df[['Glucose','BloodPressure','SkinThickness', 'Insulin','BMI']].replace(0,np.nan)
Zobaczmy jak teraz wyglądają nasze dane. W miejscu gdzie były zera mamy teraz NaN.
Sprawdźmy jak dużo jest pustych wartości w poszczególnych kolumnach.
df.isnull().sum()
Teraz możemy zastosować formułę kasującą niekompletne rekordy. Kasujemy i od razu sprawdzamy czy nie ma już braków.
df = df.dropna(how='any') df.isnull().sum()
Doskonale, mamy już czystą próbkę, sprawdźmy ile teraz mamy wierszy.
df.shape
Kolumną wynikową w naszym badaniu jest kolumna Output, która informuje czy ktoś wykazuje symptomy choroby czy nie. Zmienimy teraz wartości 1 i 0 w tej kolumnie na wartości: 'diabetis' i 'healthy'.
df['Outcome']=df['Outcome'].map({1:'diabetis', 0:'healthy'})
Sprawdzamy jak wygląda teraz nasza tabela.
df.tail(3)
Teraz, za pomocą Seaborn stworzymy duży wykres macierzowy, który przedstawi zależności pomiędzy zmiennymi egzogenicznymi.
sns.pairplot(data=df[['Pregnancies','Glucose' ,'BloodPressure','SkinThickness','Insulin','BMI','DiabetesPedigreeFunction','Age', 'Outcome']], hue='Outcome', dropna=True)
Wprawione oko badacza szybko znajdzie, które zmienne egzogeniczne mają duży wpływ na powstanie choroby.
Teraz zmniejszymy ilość parametrów aby lepiej się im przyjrzeć.
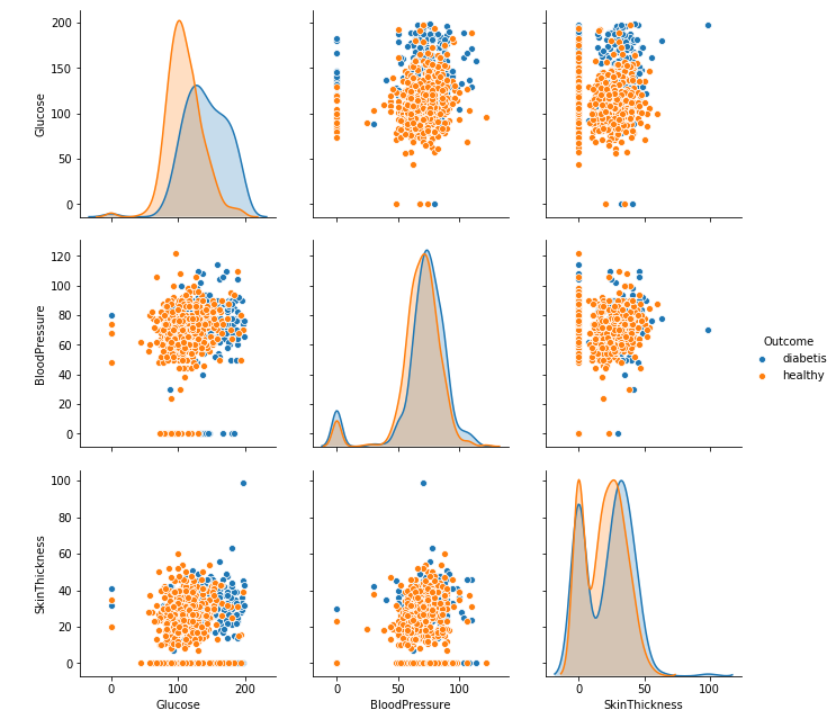
sns.pairplot(data=df[['Glucose' ,'BloodPressure','SkinThickness', 'Outcome']], hue='Outcome', dropna=True, height=3)
Wyraźnie widać, że najbardziej wpływowy parametrem określającym zachorowalność jest poziom glukozy we krwi. Widać to ponieważ niebieskie punkty wskazujące na zapadanie na cukrzycę pojawiają się wyraźnie przy przekroczeniu pewnej granicznej wartości poziomu glukozy we krwi. Super, ale chyba właśnie główny symptom tej choroby jest podwyższony poziomu glukozy we krwi. Niestety nie znam się za bardzo na medycynie. Dochodzimy do oczywistych wniosków, a przecież chodziło nam na zbadaniu jakie parametry przepowiadają zapadalność na cukrzyce.
Zbadajmy za pomocą Seaborn więc zależność innych parametrów, przy okazji zmienimy troszkę kolory i kształt znaczników na wykresie.
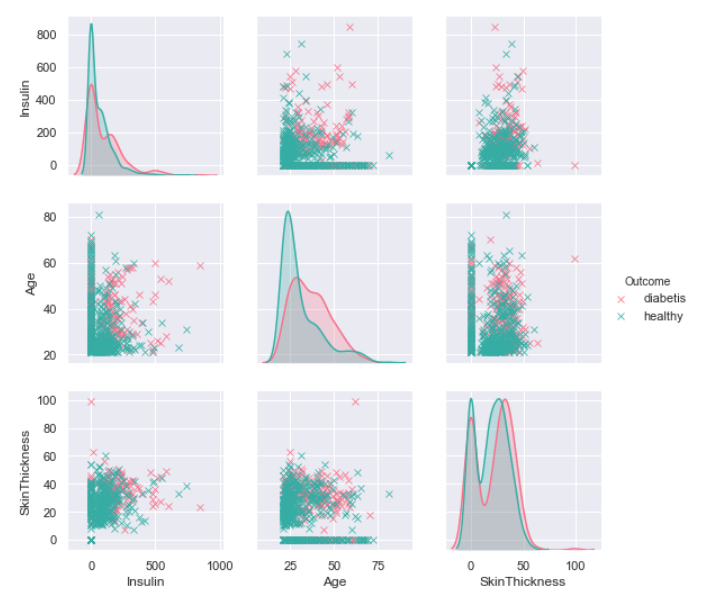
sns.pairplot(data=df[['Insulin' ,'Age','SkinThickness', 'Outcome']], hue='Outcome', dropna=True, height=2.5, palette=('husl'), markers='x')
Możemy ten sam wykres przedstawić w trochę innej formie.
g = sns.PairGrid(data=df[['Insulin' ,'Age','SkinThickness', 'Outcome']], hue='Outcome', dropna=True, height=3) g = g.map_upper(plt.scatter) g = g.map_lower(sns.kdeplot)
Widzimy wyraźnie, że na zapadalność na cukrzycę istotny wpływ ma poziom insuliny oraz wiek badanych osób. Widać to na wykresie po wyraźnych zmianach kolorów.
g = sns.pairplot(data=df[['Insulin' ,'Age','SkinThickness', 'Outcome']], hue='Outcome', dropna=True, height=3, aspect=2, palette=('cubehelix'), markers='o')
g.axes[2,1].set_xlim((0,140))
g.axes[1,2].set_xlim((0,60))